LE PROGRAMME SCIENTIFIQUE DE LIST_MAPS
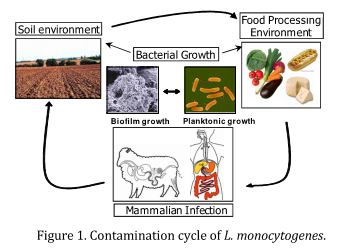
Les mécanismes permettant à Listeria monocytogenes de transiter et de persister dans des habitats très variés restent encore mal connus. Il est cependant clair que la capacité d’intégrer des signaux environnementaux et de développer une réponse appropriée aux conditions de son environnement participe au risque sanitaire associé à la présence de cette bactérie dans les environnements alimentaires.
Le cœur du projet scientifique de List_MAPS est d’intégrer la réponse de la bactérie aux conditions environnementales par une approche de biologie des systèmes et comprendre comment les conditions environnementales peuvent influencer sa capacité à générer une infection après ingestion.
Quatre dispositifs expérimentaux sont utilisés pour étudier en détail l’écologie de Listeria monocytogenes :
- Le sol et la rhizosphère (zone entourant les racines des plantes)
- Les biofilms de l’environnement agro-alimentaire
- L’aliment et les stress technologiques
- La souris comme modèle d’hôte pour l’étude du pouvoir infectieux
Le réseau étudie notamment :
- Les transcriptomes et protéomes dans ces environnements complexes
- Les mécanismes d’adaptation physiologique par des approches de génétique inverse
- Les interconnections entre la réponse au stress, la communication cellulaire et le RNome
- La réponse à l’environnement biotique
- Le lien entre la composition des matrices alimentaires et la capacité du pathogène à initier un processus infectieux
- Le développement d’un outil d’évaluation de la virulence innovant en remplacement aux modèles animaux pour cribler des collections de souches pour lier biodiversité et pouvoir infectieux
- La mise en œuvre d’un dispositif commercial pour évaluer la capacité à former des biofilms dans des conditions technologiques
- La lumière comme outil de désinfection pour le secteur agroalimentaire
ORGANISATION DES ACTIVITÉS DE RECHERCHE
Les activités de recherche de List_MAPS sont organisées en 4 ensembles de tâches combinant les approches globales (transcriptome et protéome), les approches classiques de biologie moléculaire et les caractérisations physiologiques dans les quatre environnements contrôlés:
WP1: Data collection and integration in the specific environments
Objectifs: Extraction des ARN et des protéines dans les divers environnements (tâche 1) ; construction d’une base de donnée incluant l’identification des TSS, les analyses RNA-seq et MACE (tâche 2) ; et l’intégration des données de transcriptomique et de protéomique (tâche 3).
Partenaire responsable: GenXPro
WP2: Linking environmental cues and expression of virulence
Objectifs: Évaluer l’effet des constituants de la matrice alimentaire et de la présence de chitine sur le niveau de virulence (tâche 1), exploitation des données de transcriptome pour l’analyse de l’expression du virulon en fonction des conditions environnementales (tâche 2), évaluation de la virulence d’une collection de mutants (tâche 3).
Partenaire responsable: University College Cork
WP3: Tools for evaluation of intra-specific phenotypic diversity
Objectifs: Développement et validation d’un test innovant d’évaluation de la virulence in silico en remplacement des modèles animaux (tâche 1), développement d’un kit d’évaluation de la formation de biofilm (tâche 2), analyse de la diversité d’une collection de souches avec ces outils (tâche 3).
Partenaire responsable: BioFilm Control
WP4: Systems biology approach
Objectifs: Modélisation des réseaux de régulation (tâche 1), identification de gènes cibles et approche de génétique inverse (tâche 2), optimisation du modèle grâce à la confrontation des résultats expérimentaux et des hypothèses tirées du modèle théorique (tâche 3).
Partenaire responsable: INRA
PROJETS DE RECHERCHE INDIVIDUELS
Le projet scientifique global du réseau nécessite la contribution de chacun des projets de recherche individuel. Ces projets individuels sont au cœur de la formation par la recherche des jeunes chercheurs.
![]() Ce projet est financé par les actions Marie-Sklodowska Curie du programme de recherche et d’innovation de l’Union européenne Horizon 2020 selon l’accord de subvention n°641984
Ce projet est financé par les actions Marie-Sklodowska Curie du programme de recherche et d’innovation de l’Union européenne Horizon 2020 selon l’accord de subvention n°641984